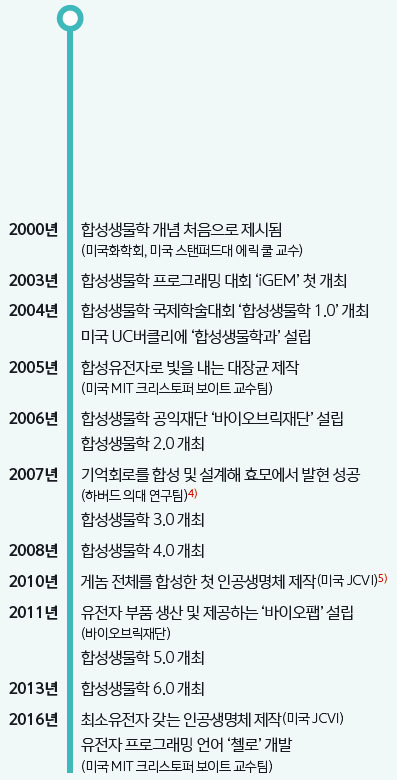
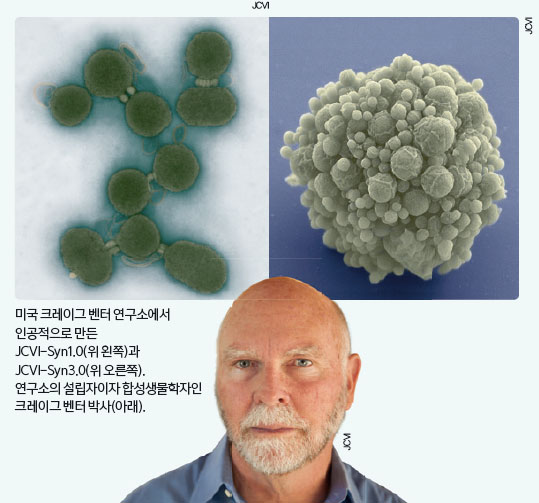
2010년 5월 ‘사이언스’에는 최초의 인공생명체가 태어났다는 충격적인 논문이 발표됐다. 미국의 합성생물학자인 크레이그 벤터 연구팀의 성과였다. 연구진은 마이코플라스마 마이코이데스라는 박테리아의 유전자 전체를 화학적으로 합성한 뒤 또다른 박테리아인 마이코플라스마 카프리콜럼의 유전자와 ‘바꿔치기’ 했다. 카프리콜럼의 ‘몸(세포)’에 마이코이데스의 ‘영혼(유전자)’이 들어간 이 생물은 놀랍게도 성장과 분열을 거듭했다. 이것이 ‘JCVI-Syn1.0’이다.
그리고 6년 뒤인 올해 3월, 자연계에는 존재하지 않는 새로운 생명체 ‘JCVI-Syn3.0’이 탄생했다1). Syn1.0에서 생존과 번식에 필수적인 최소한의 유전자만 선별해 디자인(설계)한 진정한 의미의 ‘새로운 생명체’다. 벤터 박사가 2008년 테드 강연에서 “디지털 세계에서 새로운 생명을 창조해낼 수 있다”고 공언한 지 만 8년 만에 성공한 것이다. 인간의 디자인으로 만들어진 첫 생명체, JCVI-Syn3.0(이하 Syn3.0)은 어떻게 탄생한 것일까.
유전자를 전자회로처럼 설계한다
Syn3.0을 제대로 파악하려면 탄생 배경을 먼저 알아야 한다. Syn3.0을 제작한 벤터 박사는 처음부터 합성생물학에 관심이 있었던 건 아니었다. 그가 유전자를 합성하게 된 건 ‘최소유전자’를 밝히기 위해서였다. 1995년 자신이 설립한 게놈연구소(TIGR)에서 마이코플라스마 제니탈리움의 게놈을 모두 읽어내는 데 성공한2) 벤터 박사는 4년 뒤, 제니탈리움의 유전자 중 265~300여 개는 생존에 필수적이지 않다는 연구 결과를 발표했다.3) 그는 여기서 그치지 않고 정말 최소유전자들만 가진 생명체가 생존할 수 있는지를 연구하기 시작했다.
유전자재조합 기술을 이용해 생존에 필수적이지 않은 유전자의 기능을 없애버리면 쉽게 확인할 수 있을 것 같지만, 생명체란 그리 간단하지 않다. 유전자 연구를 하는 데 가장 큰 걸림돌은 생물이 지닌 복잡성이다. 유전자는 마치 그물망처럼 서로 얽히고설켜 하나의 요소만 떼어내도 그물망 전체가 기능을 하지 못한다. 그러니 자연계에 존재하는 게놈에서 유전자를 하나씩 없애는 방식으로는 최소유전자를 확인할 수 없다. 반대로, 생명체의 설계단계부터 다시 시작하는 게 낫다. 이런 배경에서 나온 것이 합성생물학이다.
합성생물학에서는 유전자를 하나의 부품으로 본다. 마치 레고의 블록처럼. 만약 합성생물학의 관점대로 유전자가 레고의 블록 같은 존재라면, 생물의 복잡성 문제를 좀 더 간단히 풀 수 있다. 레고는 하나의 조각을 떼어내도 전체 형태(기능)를 유지하는 데 문제가 없다. 장난감을 지탱하는 필수적인 조각들이 사라질 때만 형태가 무너진다. 벤터 박사는 최소유전자를 확인하기 위해 레고(유전자)를 쌓아나가는 방법을 선택했다.
이렇게 유전자를 하나의 레고 조각으로 만드는 것을 합성생물학에서는 ‘유전자의 부품화’라고 하고 레고를 쌓아나가는 것을 유전자를 ‘설계’한다고 말한다. 전자회로에도 트랜지스터, 다이오드, 저항 등 부품이 존재하는 것처럼 합성생물학에도 ‘프로모터 서열’, ‘핵심 유전자 서열’, ‘리보솜결합 서열’, ‘터미네이터 서열’ 등의 유전자 부품이 있다. 합성생물학자들은 유전자 부품의 서열 정보를 이용해 전자회로를 구성하듯 생체회로를 설계한다.
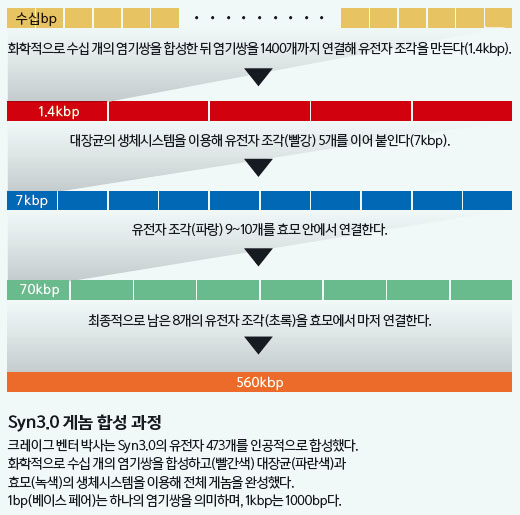
DNA 읽기, 쓰기, 편집 기술이 필요해
여기엔 레고 조립보다는 조금 더 어렵고 복잡한 합성생물학 기술이 필요하다. 크게 ‘DNA 읽기’, ‘쓰기’, ‘편집’ 세 가지가 있다. DNA 읽기 기술은 말 그대로 DNA의 염기서열을 읽어내는 기술로, DNA 시퀀싱이라고도 한다. 이 기술은 현재 인간 게놈 전체를 읽어내는 데 2~3일이면 충분할 정도로 많이 발전한 상태다. 미국 국립보건원(NIH)이 올해 2월에 밝힌 인간 게놈 시퀀싱 비용을 보면 2001년 1억달러(약 1151억 원)에 달하던 것이 2015년에는 1000달러(약 115만 원)로 10만분의 1 가량으로 줄었다. 그만큼 시퀀싱 기술은 완성에 가까운 상태다.
아직 발전이 좀 더 필요한 기술은 ‘DNA 쓰기’다. 원하는 유전자를 실험실 조건에서 인위적으로 만들어내는 기술이다. 유전자를 이루는 염기서열 A(아데닌), G(구아닌), T(티민), C(시토신)은 모두 화학물질이기 때문에 화학적으로 합성할 수 있다. 읽기 기술은 2000년 초반부터 지수곡선 형태로 급격히 발전한 반면, 쓰기 기술은 합성할 수 있는 염기쌍 수가 여전히 1차 함수 형태로만 늘고 있다.
문제는 정확성이다. 방두희 연세대 화학과 교수는 “현재 화학적 합성으로는 염기 100개 마다 1개 꼴로 오류가 발생한다”며 “수백 개 이상의 염기로 원하는 유전자를 한번에 합성하는 건 불가능하다”고 말했다. 이런 이유로 수십 개의 염기쌍을 화학적으로 합성한 뒤, 이 조각들을 연결해 큰 조각을 만든다. 이를 ‘유전자 조립(gene assembly)’이라고 부른다.
조립 방법에는 여러 가지가 있는데, 최근 가장 주목을 받는 건 효모를 이용한 방법이다. 벤터 박사 역시 효모를 이용해 유전자를 연결했다. 방 교수는 “화학적 합성에서 발생하는 오류율을 줄이는 연구가 진일보한다면 짧은 시간 안에 저렴한 가격으로 유전자를 합성하는 시대도 곧 올 것”이라고 말했다.
과학자도 탐내는 생명체
이 기술들의 합심으로 만들어진 Syn3.0이 학계의 주목을 받고 있는 건, 처음으로 한 생명체의 게놈을 처음부터 끝까지 설계한 사례기 때문이다. 합성생물학이 처음 등장한 2000년대 초반부터 당 분해 같은 하나의 기능을 하는 유전자 모음인 오페론 수준의 설계는 있어왔지만 유전자 전체, 게놈 단위의 설계는 이번 연구가 처음이다. 이승구 한국생명공학연구원 바이오합성연구센터장은 이를 ‘서고 정리’에 비유했다. 서고에 어지럽게 널려 있는 책들 중 논문을 쓰는 데 필요한 책을 선별하고, 한 편의 글을 쓰기 위해 필요한 책을 순서대로 정리한 셈이다. 만약 기존 서고에 새로운 책을 추가한다면, 별로 눈에 띄지 않아 새 책의 역할을 확인하기 어려울 것이다. 하지만 정리된 서고에 놓은 새 책은 쉽게 역할을 파악할 수 있다.
예컨대 에탄올을 생산하는 합성유전자를 대장균에 넣었다고 해보자. 대장균(정리 전 서고)이 에탄올을 생산하더라도, 우리가 환경과 유전자를 제어했기 때문인지 혹은 예상치 못한 다른 변수 때문인지 확인할 방법이 없다. 이런 점에서 유전자와 변수를 최소화한 Syn3.0(정리된 서고)은 합성유전자의 기능을 확인하고 구현하는 데 아주 적합한 생물이다. 이 센터장은 “살 수만 있다면 Syn3.0은 직접 구입해서 실험해 보고 싶을 정도로 탐나는 생명체”라며 “Syn3.0은 합성생물학을 발전시키는 견인차 역할을 할 것”이라고 전망했다.