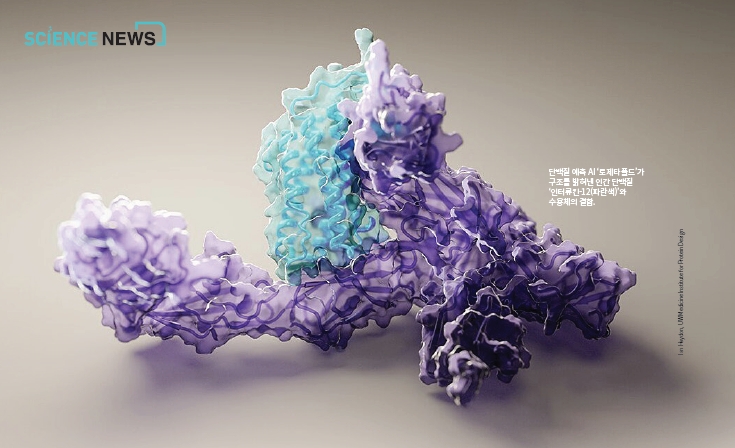
수십여 년 동안 구조생물학자들은 단백질의 3차원(3D) 구조를 정확히 예측하기 위해 고군분투해왔다. 단백질 구조는 단백질 기능과 직결되기 때문에 구조를 알아내면 연구자들이 신약 개발이나 생리적인 특성을 파악하는 데 도움이 될 수 있다. 그런데 인공지능(AI)의 등장으로 단백질 구조 예측 연구의 판도가 바뀌었다. 딥마인드가 개발한 AI 알파폴드2(AlphaFold2)는 지난해 12월 ‘제14회 단백질 구조예측 대회(CASP14)’에서 대회 사상 최고점을 기록했다. 하지만 알파폴드2의 소스 코드가 공개되지 않아 과학자들이 연구에 활용하진 못했다.
7월 15일 딥마인드 연구팀은 알파폴드2의 소스 코드와 설계 방법을 각각 코드 공유 플랫폼 ‘깃허브’와 국제학술지 ‘네이처’에 공개했다. doi: 10.1038/s41586-021-03819-2 공개한 자료에는 인간 유전체에 기록된 단백질 2만여 개와 생물 20종의 단백질 구조가 포함됐다. 이번에 공개된 알파폴드2의 소스 코드는 알고리즘을 간소화해 CASP14에 참가했을 때보다 예측 시간이 16배 빨라졌다.
같은 날, 알파폴드2에 영감을 받아 탄생한 미국 워싱턴대 베이커랩의 단백질 구조예측 AI ‘로제타폴드(RoseTTAFold)’의 설계 방법도 국제학술지 ‘사이언스’에 발표됐다. doi: 10.1126/science.abj8754 로제타폴드의 소스 코드는 이미 6월 깃허브에 공개됐다. 로제타폴드도 알파폴드2와 우열을 가릴 수 없을 정도로 빠르며, 성능도 뛰어났다.
두 AI는 모두 아미노산의 배열과 단백질의 구조를 학습해 새로운 아미노산 배열에서 단백질의 구조를 2차원 형태로 도출한 다음 3차원 구조를 만들어 낸다. 로제타폴드 개발에 참여한 백민경 베이커랩 연구원은 e메일 인터뷰에서 “로제타폴드가 알파폴드2와 구별되는 점은 생성된 3차원 구조 및 다시 2차원 구조, 서열과 끊임없이 상호작용해 최종적인 3차원 구조를 생성하는 것”이라며 “알파폴드2는 이 상호작용이 약하고 3차원 구조로 처음부터 시스템을 다시 수행하는 재사용 과정을 거친다. 두 AI가 때때로 속도 차이를 보이는 이유가 바로 이 때문”이라고 말했다.
단백질 구조 예측 AI는 이미 다양한 연구에 쓰이고 있다. 미국 샌프란시스코 캘리포니아대 생화학 및 생물물리학부팀은 알파폴드2와 극저온전자현미경의 관찰 결과를 결합해 사스코로나바이러스-2가 인체에 침입할 때 이용하는 비구조단백질2(Nsp2) 구조를 밝혀냈다. doi: 10.1101/2021.05.10.443524 백 연구원은 “로제타폴드와 알파폴드2 모두 공개된 단백질 서열과 구조를 토대로 만들어졌기 때문에 연구자들이 활용할 수 있도록 하는 게 당연하다”며 “AI는 단백질 구조 예측 분야의 돌파구이기 때문에 많은 연구자들이 사용해 생물학, 의학 등이 더욱 발전하길 기대한다”고 말했다.