“앞으로 반도체 집적회로의 성능은 18개월마다 2배씩 증가할 것이다.”
인텔의 공동창립자인 고든 무어가 1965년 4월 19일자 ‘일렉트로닉스(Electronics)’ 잡지에 쓴 ‘집적회로에 더 많은 부품 집적하기(Cramming More Compo nents onto Integrated Circuits)’라는 기고문에 담긴 내용이다. 정확하게는 무어가 이런 얘기를 한 것이 아니라, 카버 미드 캘리포니아공대 교수가 글의 핵심 내용을 이렇게 요약하며 ‘무어의 법칙(Moore’s Law)’으로 부르면서 굳어졌다.
무어의 법칙은 같은 면적에서 메모리의 용량이, 또 중앙처리장치(CPU)의 속도가 2배씩 향상된다는 의미이기도 하다. 2000년대 초반까지만 해도 무어의 법칙은 실제 ‘법칙’처럼 잘 맞아 떨어지는 듯 보였다. 하지만 점점 집적회로가 작아지면서 발열 문제 등 기술적 한계에 봉착했다.
특히 인공지능(AI), 빅데이터, 사물인터넷(IoT) 등 새로운 산업이 등장하면서 지금보다 훨씬 작고 효율적인 메모리의 필요성이 더욱 커지고 있다. 과학학술지 ‘네이처’는 2016년 8월 31일 “전 세계는 데이터 위기에 직면해 있다”며 “2020년에는 천문학적인 양의 이미지와 유튜브 동영상 등으로 필요한 메모리 용량이 약 44조 기가바이트(GB)에 이를 것”이라고 예측했다. 이는 2013년에 소비된 메모리 용량의 10배에 달하는 크기다.
전문가들은 수년 째 지금과는 전혀 다른 새로운 패러다임의 메모리가 필요하다고 지적하고 있다. 실리콘메모리를 대신할 완전히 새로운 메모리가 있을까.
제1 후보는 ‘DNA 메모리’
첫 번째 후보는 ‘DNA 메모리’다. DNA 메모리는 DNA를 구성하는 아데닌(A), 구아닌(G), 티민(T), 시토신(C)등 염기 4개에 이진수로 표현된 정보를 저장하는 메모리다. 가령 A는 ‘00’, G는 ‘01’, T는 ‘10’, C는 ‘11’로 표현할 수 있다. AG는 ‘0001’이 되는 셈이다.
기존의 기억장치는 0과 1, 2개의 비트로 정보를 저장했지만, DNA 메모리는 4비트로 정보를 저장할 수 있기 때문에 메모리의 밀도가 대폭 높아진다. 기존 하드디스크의 정보 기억 밀도는 1mm2 당 1010바이트인 반면, DNA의 저장 밀도는 1mm2 당 1018바이트 정도로, 1억 배 이상 높다. 더구나 DNA는 현재 메모리 장치에 사용되는 실리콘보다 외부 자극에 강하기 때문에 장기 저장이 가능하다.
DNA를 메모리로 처음 활용한 건 2012년이었다. DNA 합성 연구로 유명한 조지 처치 미국 하버드대 교수팀은 65만8750바이트 분량의 책 정보를 DNA에 저장하는 데 성공했다. 처치 교수는 자신이 쓴 ‘재생: 어떻게 합성생물학은 자연과 우리를 새롭게 만들었는가(Regenesis: How Synthetic Biology Will Reinvent Nature and Ourselves)’를 DNA에 저장하면서 일종의 전자책을 다시 출판한 셈이 됐다. 여기에는 5만3000여 개의 단어와 사진 11장이 포함됐다.
유럽생물정보학연구소(EBI)는 셰익스피어의 시 154편, DNA 이중나선 구조를 밝혀낸 프랜시스 크릭과 제임스 왓슨의 논문 등 65만 바이트에 해당하는 정보를 DNA에 담았다가 이를 다시 100% 복원하는 데 성공해 2013년 ‘네이처’에 발표했다.
대장균 유전체에 동영상 저장 뒤 복원
처치 교수팀은 살아있는 대장균의 유전체에 사진과 동영상 파일을 저장하고 복원하는 전과정을 성공시켰다. 말이 달리는 모습을 연속적으로 촬영한 사진을 DNA에 저장해 동영상으로 만들어 지난해 7월 13일 ‘네이처’에 공개했다.
연구진은 2진법으로 표현된 사진의 픽셀 정보를 염기서열로 바꿨다. ‘00’은 C, ‘01’은 T, ‘10’은 A, ‘11’은 G로 치환한 뒤, 사진 정보에 해당하는 DNA 가닥을 합성했다. 예를 들어 픽셀 정보가 ‘00011011’로 치환되면 ‘CTAG’라는 DNA 가닥을 만들었다. 연구진은 이렇게 만든 DNA 가닥을 대장균에 넣었고, 대장균에 존재하는 면역 시스템인 ‘크리스퍼(CRISPR)’에 의해 외부 DNA가 대장균의 유전체에 쉽게 삽입됐다.
대장균은 외부 바이러스가 들어오면 바이러스 유전자의 일부를 잘라 크리스퍼 서열에 삽입한다. 그 바이러스가 다시 들어오면 크리스퍼는 저장해 놓은 바이러스 유전자를 이용해 다시 들어온 바이러스의 유전자를 잘라 활동하지 못하게 한다. 이 원리를 이용한 것이 유전자가위 ‘크리스퍼-캐스9’이다. 연구진이 넣어준 외부 DNA를 적으로 인식한 대장균이 자신의 크리스퍼 서열에 이를 삽입했고, 이렇게 저장된 DNA 정보는 90%의 정확도로 복원됐다.

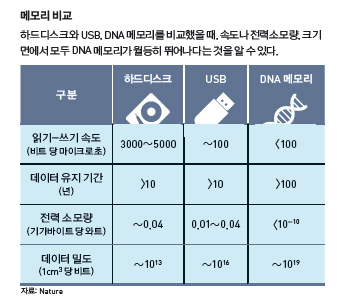
하지만 DNA 메모리가 경쟁력을 갖기 위해서는 관련 생물학 연구가 뒷받침 돼야 한다. 아직은 DNA 합성에 드는 비용이 높아 가격경쟁력이 떨어진다. 현재 DNA 생성 속도는 초당 400바이트로, 200MB에 해당하는 DNA를 합성하기 위해서는 약 80만 달러(약 9억 원)가 든다.
방두희 연세대 화학과 교수는 “DNA 시퀀싱(염기서열 분석) 기술도 처음에는 100억 원을 웃돌며 비용 문제로 경쟁력이 없다고 평가 받았지만 2000년대 중반부터 비용이 점점 낮아져 2012년에는 1000만 원대로 떨어졌다”며 “DNA 합성 역시 기술이 좀 더 성숙하면 충분히 가격을 낮출 수 있을 것”이라고 말했다.
최근에는 마이크로소프트(MS)등 글로벌 IT 기업이 DNA 메모리 사업에 뛰어들면서 적정한 시장 가격이 형성될 것이라는 전망도 나온다. 마이크로소프트는 2016년 7월 200MB에 해당하는 영상을 DNA에 저장하는 데 성공했으며, DNA 기반의 데이터센터를 10년 안에 구축하겠다는 목표를 지난해 5월 공식적으로 발표했다.
마이크로소프트 연구진은 미국 매사추세츠공대(MIT)가 발행하는 과학전문지 ‘테크놀로지 리뷰’ 2017년 5월 22일자 온라인판에 “세상의 모든 데이터를 DNA에 기록한다고 해도 자동차 트렁크에 들어갈 정도로(메모리 크기가) 작을 것”이라며 “우리가 개발하는 데이터 장치 역시 프린터 크기 정도”라고 말했다.
USB 크기에 전 세계 영화 저장, 원자 메모리
DNA보다 더 작은 메모리도 등장했다. 원자 하나에 정보를 입력하는 ‘원자 메모리’다. 샌더 오테 네덜란드 델프트공대 교수팀은 원자와 원자 사이의 극도로 작은 공간에 정보를 입력하는 방식의 메모리를 구상해 2016년 7월 ‘네이처 나노테크놀로지’에 발표했다. 연구진은 구리 기판 위에 염소 원자를 배열한 뒤, 원자 사이에 전류가 흐르면 ‘1’, 흐르지 않으면 ‘0’으로 정보를 입력했다.
연구진은 수 nm(나노미터·1nm는 10억 분의 1m)의 아주 얇은 탐침을 가진 주사터널링현미경(STM)을 이용해 원자의 위치를 조정했다. 가령 ‘01010100’이 T를 의미한다면, 염소 원자를 ‘있고, 없고, 있고, 없고, 있고, 없고, 있고, 있고’ 식으로 배열한다. 마치 물(전류)이 돌(염소 원자)을 피해 흐르는 것처럼, 염소 원자가 없는 곳으로 전류가 흐르는 원리를 이용한 것이다.
연구진은 미국의 천재적인 이론물리학자 리처드 파인만이 1959년 미국물리학회 정기총회에서 강연한 ‘바닥에는 여지가 많다(There’s Plenty of Room at the Bottom)’의 내용을 원자에 입력했다. 이렇게 하면 가로, 세로 6.5cm 크기의 정사각형 칩에 500TB(테라바이트·1TB는 1조 바이트)의 정보를 입력할 수 있다.
국내에서도 원자를 활용한 메모리 연구가 한창이다. 기초과학연구원(IBS) 양자나노과학연구단은 홀뮴 원자의 스핀을 이용해 원자 메모리를 만드는 데 성공했다. 현재 사용되는 실리콘 메모리는 1비트의 정보를 저장하기 위해서 약 10만 개의 원자가 필요한 반면, 홀뮴원자 메모리는 원자 하나로 가능하다.
홀뮴은 자기적, 전기적 성질이 강한 희토류 금속 중 하나로 자연 상태에서 위와 아래, 두 가지 스핀을 갖는다. 이 성질을 이용해 0과 1을 스핀의 방향에 각각 대입해 정보를 저장한다. 주사터널링현미경(STM)을 이용해 스핀의 방향을 조정할 수도 있다.

홀뮴 원자 메모리가 특별한 이유는 홀뮴 원자들 사이의 거리가 1nm로 아주 가까워도 서로 영향을 주지 않는다는 점이다. 즉 그만큼 원자의 집적도를 높여 메모리를 작게 만들 수 있다는 의미다.
최태영 IBS 양자나노과학연구단 연구위원은 “지금까지 상영된 전 세계 모든 영화를 USB 메모리 한 개 크기의 칩에 담고도 남을 만큼 집적도가 높다”며 “앞으로 메모리 산업은 누가 더 작은 칩에, 더 안정적으로 정보를 담는지에 따라 판도가 좌우될 것”이라고 말했다.